|
|
本帖最后由 kexue 于 2024-4-21 16:43 编辑
生存分析是一种统计方法,用于预测事件(如死亡、疾病复发等)的发生时间。Kaplan-Meier曲线(KM曲线)是生存分析中常用的一种工具,用于估计在一段时间内的生存率.
一、KM曲线的主要步骤和特点包括:
1)时间排序:首先将所有参与者按照发生事件的时间排序。
2)计算生存率:对于每一个独特的时间点,计算生存率。生存率是指从研究开始到特定时间点,尚未发生特定事件的参与者比例。
3)绘制曲线:以时间为横轴,生存率为纵轴绘制曲线。每当发生事件时,曲线向下跳跃,大小与该时间点发生事件的比例成正比。
4)处理删失数据:对于那些在研究结束前未发生事件的参与者(右侧删失),他们将被纳入到最后一个已知时间点的生存率计算中,但在此之后不再影响生存率的计算。
二、如何绘制KM曲线
1)基本代码
- library("survival")
- library("survminer")
- surv_fit <- survfit(Surv(time/365, status) ~ 1, data = lung) # 计算生存曲线 这里用的lung数据集是R里自带的
- surv_fit_sex <- survfit(Surv(time/365, status) ~ sex, data = lung) # 根据性别分层,计算生存曲线
- ggsurvplot(surv_fit_sex) # 制作Kaplan_Meier曲线, 这里是最简单的KM曲线
生成的图片如下:
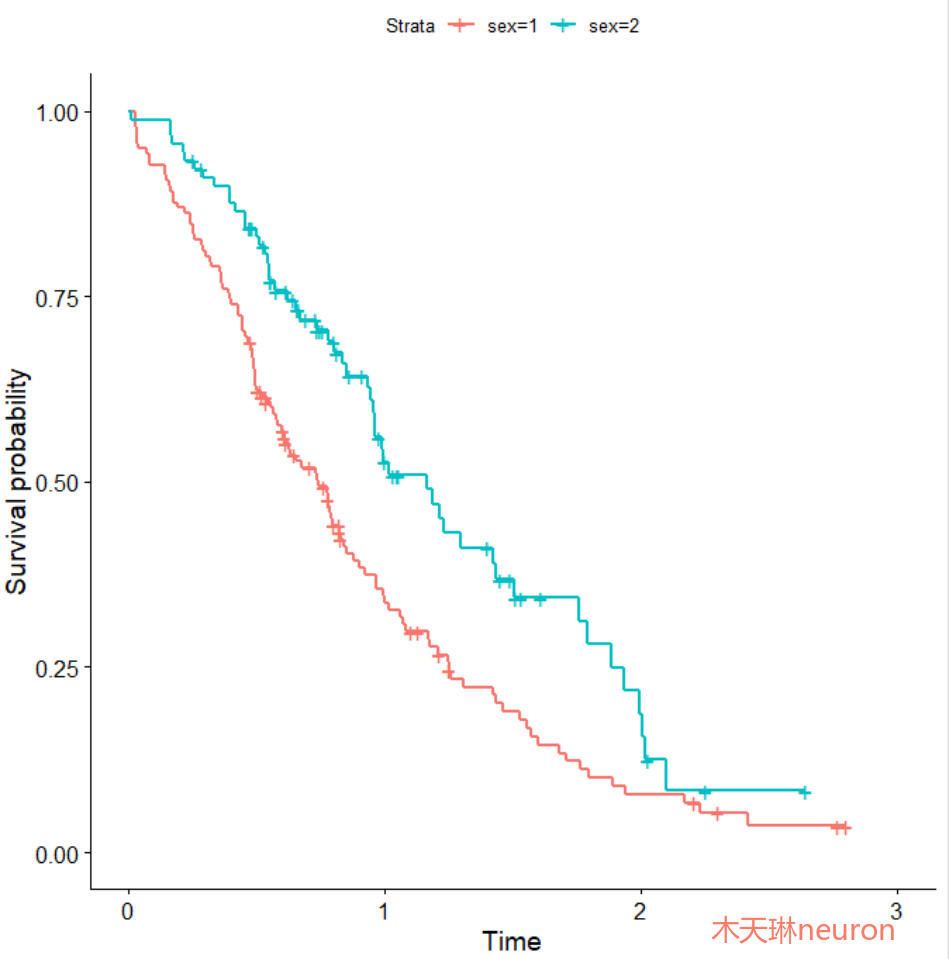
下面介绍的是如何让KM曲线更美观
2)
- ggsurvplot(surv_fit_sex,
- pval = T, #显示P值
- conf.int = TRUE, #显示置信区间
- conf.int.style='ribbon', #置信区间风格
- linetype = "strata", #根据性别分组
- surv.median.line = "hv", #显示中位生存期
- ggtheme = theme_classic(), #设置ggplot2主题
- legend.labs=c('F','M'), #改变图例标签
- legend.title='Sex', #图例标题
- legend = c(0.80,0.88), #指定图例位置
- palette = c("steelblue", "orange3")) #指定KM曲线颜色
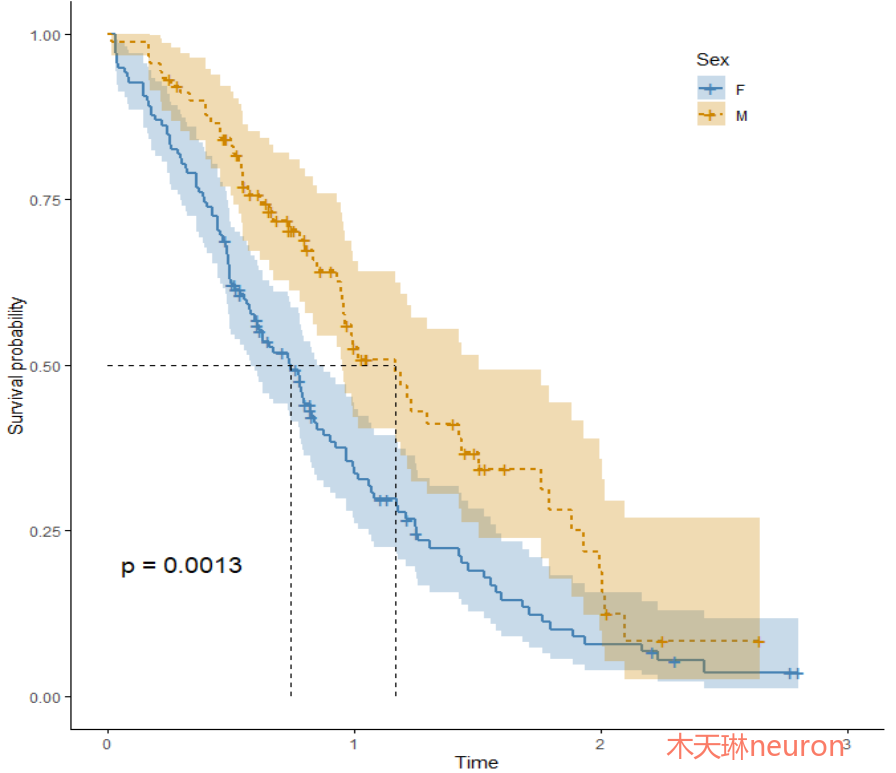
3)增加风险表格
- ggsurvplot(surv_fit_sex,
- pval = TRUE, #显示P值
- conf.int = TRUE, #显示置信区间
- risk.table = TRUE, #加风险表格
- linetype = "strata", #根据性别分组
- legend.title = "Sex", #图例标题
- surv.median.line = "hv", #显示中位生存期
- ggtheme = theme_minimal(), #设置图片主题
- palette = c("steelblue", "orange3")) #指定KM曲线颜色

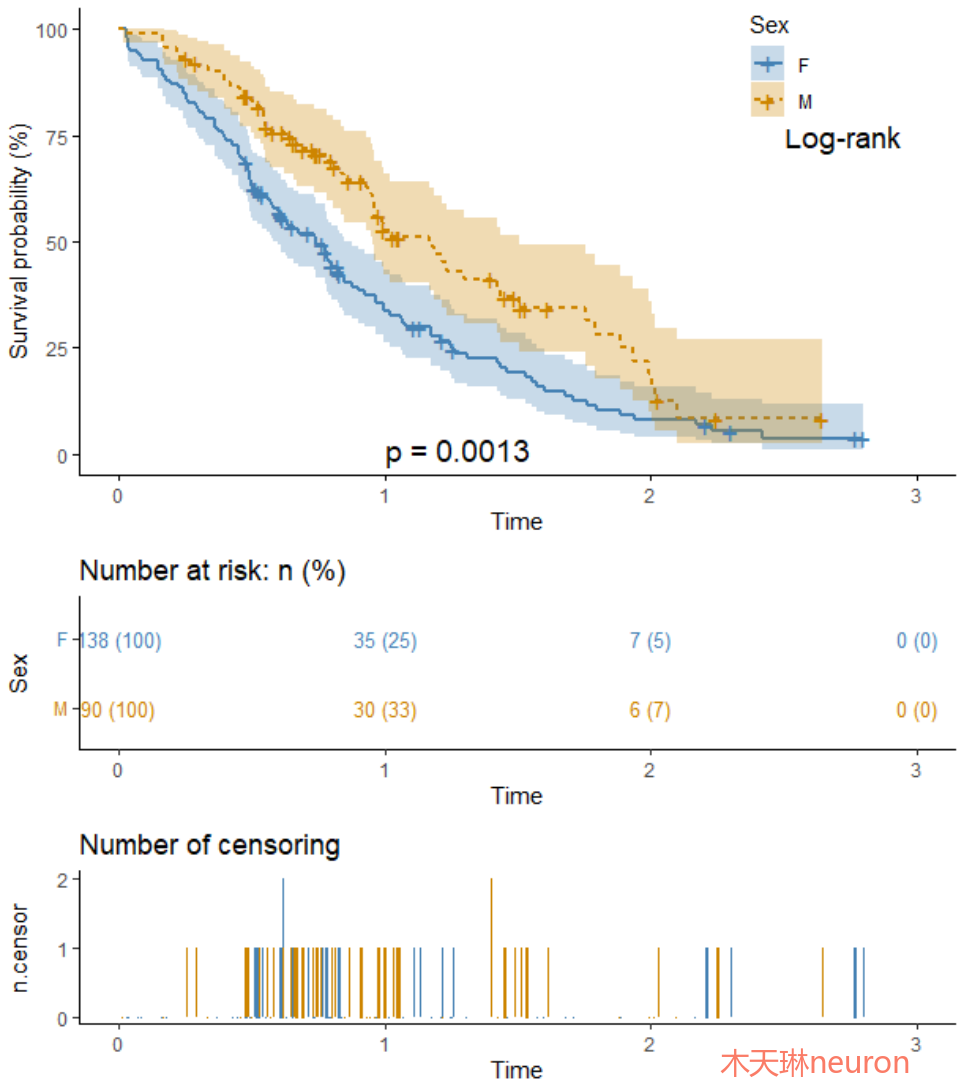
展示了在每个时间点上的风险人数(Number at risk)。这表示在该时间点还未发生事件(如死亡或失访)的受试者数量。
4)显示了每个时间点未发生事件而退出研究的受试者数量
- ggsurvplot(surv_fit_sex,
- xlab='Time', #设置X轴名
- pval = T, #显示P值
- pval.size=5, #P值字体大小
- pval.coord=c(1,1), #P值位置
- pval.method =T, #显示P值检验方法
- pval.method.coord=c(2.5, 75), #检验方法位置
- conf.int = TRUE, #显示置信区间
- conf.int.style='ribbon', #设置置信区间风格
- linetype = "strata", #根据性别设置曲线类型
- ggtheme = theme_classic(), #设置图片主题
- legend.labs=c('F','M'), #图例标签名字
- legend.title='Sex', #图例名字
- legend = c(0.80,0.88), #指定图例位置
- fontsize=3.5, #风险表字体大小
- ncensor.plot=T, #展示随访过程中不同时间点死亡和删失情况
- risk.table = 'abs_pct' , #在下面添加风险表展示绝对数和百分比
- risk.table.col='strata', #根据数据分组为风险表添加颜色
- tables.height=0.25, #下面2个表高度占比
- palette = c("steelblue", "orange3"),
- fun='pct') ##survival probability in percentage./生存率
- #fun='cumhaz',##plots the cumulative hazard function (f(y) = -log(y)),#累计风险
- #fun='event', #plots cumulative events (f(y) = 1-y), ##累计死亡人数
三、KM曲线的优点:
1)直观:KM曲线非常直观地展示了随时间变化的生存率。
2)不需要假设生存时间分布:KM方法不需要对数据的生存时间分布做出任何假设,使其适用于各种类型的生存时间数据。
3)处理删失数据:KM方法能够处理右侧删失数据,这在临床试验和生存分析中非常常见。
KM曲线在医学研究、生物统计学以及其他需要生存或事件时间分析的领域中非常有用。它们提供了一种简单而直观的方法来展示和比较不同组的生存情况。
来源:木天sy 木天琳neuron
|
|